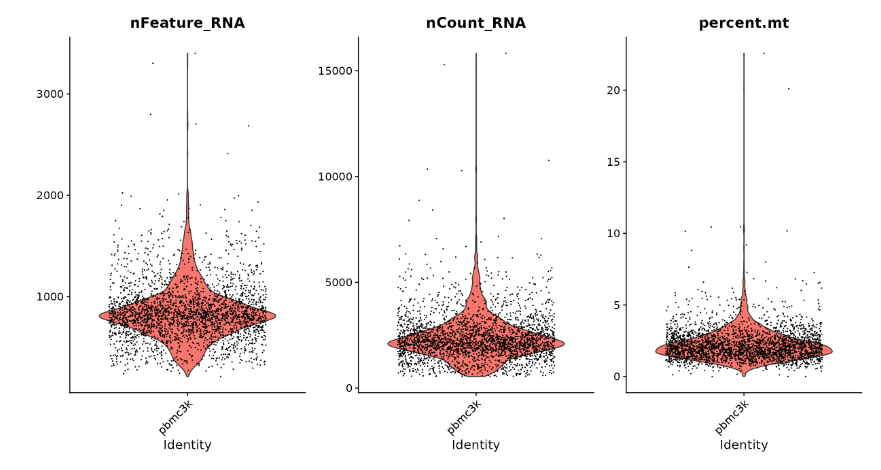
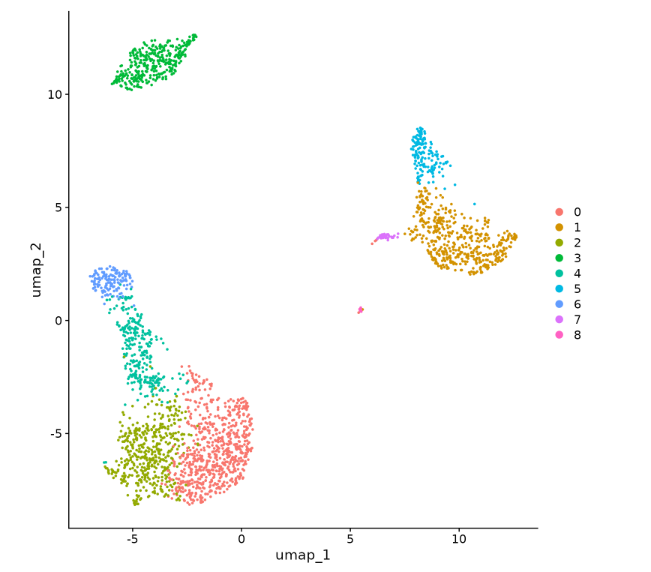
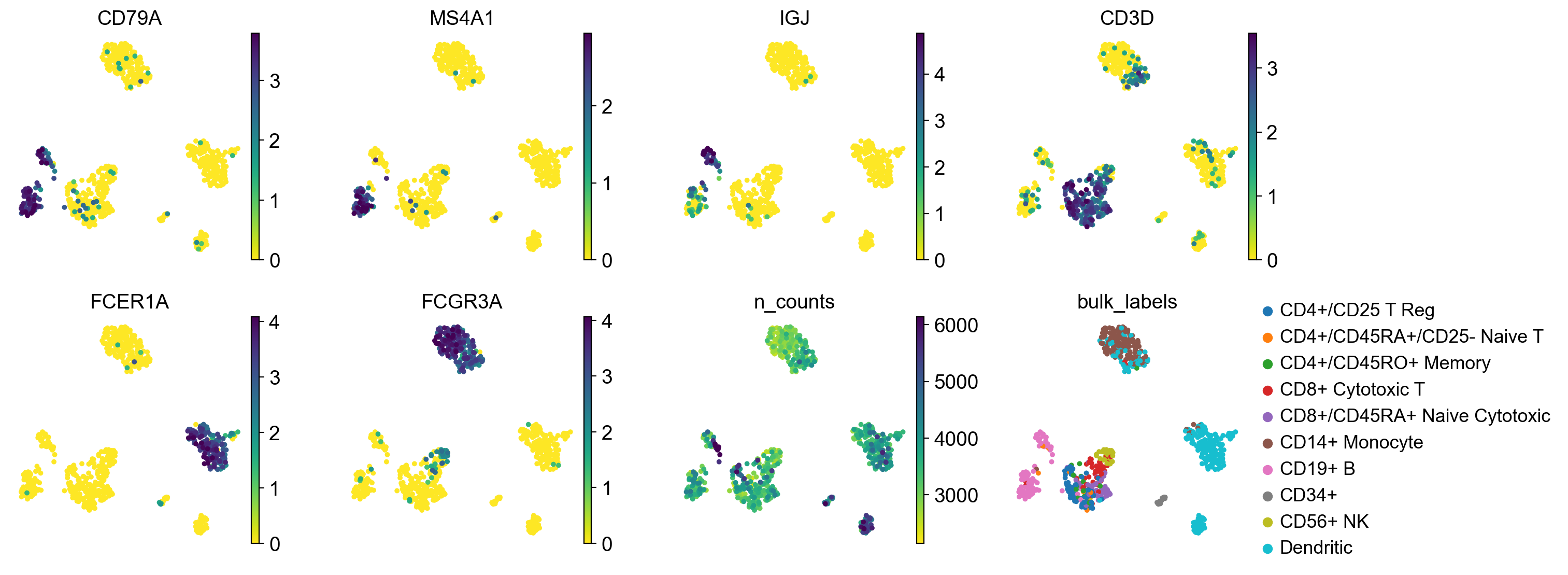
随着单细胞测序技术的快速发展,海量的单细胞数据被生成。有效的可视化能够帮助科研人员深入理解数据背后的生物学意义,是研究中至关重要的一环。通过对不同细胞亚群的可视化分析,我们能够揭示群体间的异质性和特征分布,发现关键的生物学模式和潜在调控机制。
让生信分析更简单高效,点击图片访问【生信圆桌】获取生信云服务器;下单填写优惠码 tebteb 立省200元
单细胞数据可视化工具介绍
1.Seurat
Seurat 是一种广泛使用的 R 语言工具,专门用于单细胞 RNA-seq 数据分析与可视化。Seurat 提供了多种降维和聚类算法,能够生成多样化的可视化图表,包括 UMAP、t-SNE、FeaturePlot、DotPlot 等。其丰富的功能集成了数据预处理、细胞聚类、数据整合、差异表达分析以及可视化,适用于从单细胞转录组到多组学数据的多种类型整合。
2.Scanpy
Scanpy 是基于 Python 的单细胞数据分析工具,以其高效的内存处理和丰富的绘图功能而广受欢迎。Scanpy 能够处理大型单细胞数据集,提供了包括 PCA、t-SNE、UMAP 等在内的多种降维方法。它支持与 AnnData 的无缝对接,能够生成细胞类型标注、表达热图等多种图表,特别适合在云端和分布式环境中运行。
3.其他工具
Cell Ranger:由 10x Genomics 开发的自动化单细胞 RNA-seq 数据处理工具,包含基础的细胞聚类、差异表达分析和基础可视化功能。
Louvain 和 Leiden 聚类算法:常用于大规模单细胞数据的聚类分析,以生成具有生物学意义的细胞簇。
单细胞数据可视化的图表类型与解释
1.降维图(UMAP 和 t-SNE)
UMAP(Uniform Manifold Approximation and Projection)和 t-SNE(t-Distributed Stochastic Neighbor Embedding)是单细胞数据常用的降维方法,通过将高维数据投射到二维或三维空间中展示细胞的分布情况。UMAP 和 t-SNE 图常用于显示细胞亚群的聚类结果。图中每个点代表一个细胞,颜色用于区分不同的细胞类型或状态,帮助识别群体异质性和相似的细胞群。
2.FeaturePlot 和 DotPlot
FeaturePlot 是一种用于展示特定基因在细胞群中的表达模式的图表,可以展示某基因在不同细胞类型中的分布。DotPlot 则使用点的大小和颜色来表示基因的表达水平和细胞比例,非常适合展示多个基因在不同群体间的表达差异。
3.热图(Heatmap)
热图通过颜色强度展示基因表达水平,通常用于差异基因的聚类分析。它适合对多个基因和细胞群进行比较,常用在差异表达基因分析和细胞群的标注中。
4.轨迹图(Trajectory Plot)
Trajectory Plot 用于展示细胞随时间或分化路径上的动态变化,是了解细胞发育过程的理想工具。通过轨迹图,科研人员能够识别发育阶段、分化路径及关键转录因子的变化。
单细胞数据可视化的展示方式和报告生成
1.高质量的图表展示
科研报告或发表文章时,图表应简洁清晰。推荐对颜色、尺寸等进行标准化,使图表易于理解。例如,UMAP 和 t-SNE 图中的细胞簇可以使用相对一致的调色板,以便读者快速识别主要群体。
2.整合式报告生成
Seurat 和 Scanpy 等工具支持将多种可视化集成到统一报告中。可以使用 RMarkdown 或 Jupyter Notebook 来生成包含分析流程、图表和解释的整合报告,方便展示每个数据分析步骤及其结果。
3.交互式可视化报告
为了进一步提高可视化效果,可使用 Shiny(R)或 Dash(Python)构建交互式报告,允许读者实时调节参数,探索数据特性。这种方式适合共享给更广泛的用户群体,帮助他们更深入地理解数据。
应用与前景
随着单细胞技术的广泛应用,可视化技术也在不断进步。将可视化结果生成交互式报告,结合云计算环境和多组学整合,未来的单细胞数据分析将更加高效和多元化,有助于更广泛的精准医学和细胞生物学研究。