引言:非科班医学生也能轻松掌握生信服务器搭建
随着单细胞测序(Single-cell RNA-seq)技术的快速发展,越来越多医学生开始接触生物信息学(生信)分析工具。Cell Ranger,作为10x Genomics推出的核心分析软件,几乎成为单细胞数据处理的“标配”。然而,对于非计算机科班出身的医学生来说,部署一台能够顺利运行Cell Ranger的生信服务器,常常像是一座难以逾越的“技术高墙”。
其实,只要选对服务器配置,掌握基本搭建步骤,即使是完全没有服务器经验的医学生,也能快速上手,搭建出一套稳定高效的生信分析环境。
本文将手把手教你,如何从零开始部署一个可以流畅运行Cell Ranger的生信服务器,助力你的科研项目高效推进!
Cell Ranger运行环境对服务器的基本要求
在正式搭建之前,首先需要了解Cell Ranger对服务器硬件的基本要求,以避免后期因性能不足导致分析失败或崩溃。
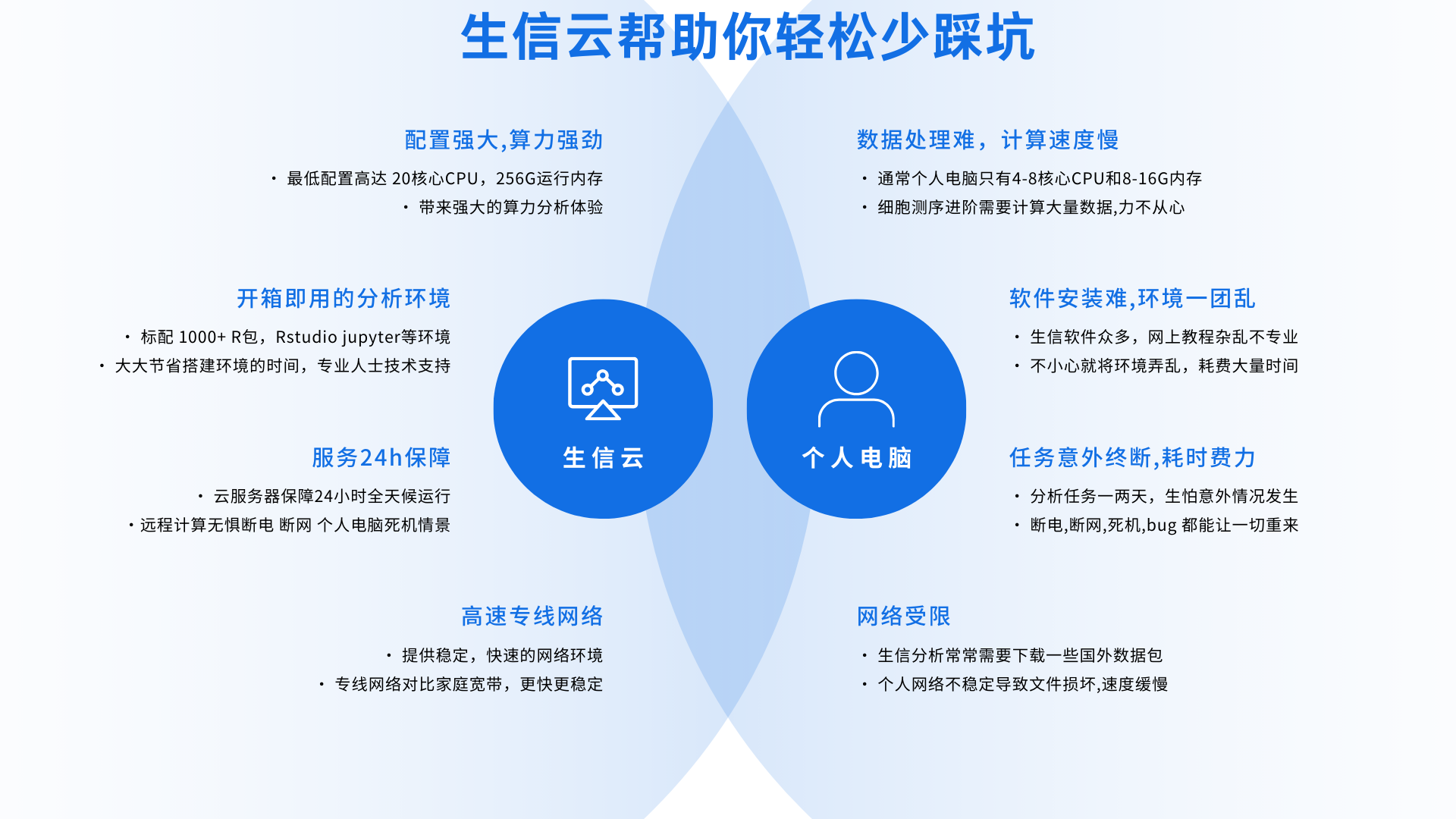
内存(RAM)要求:决定分析成败的关键
Cell Ranger在单细胞数据分析中,对内存的需求非常高。官方推荐服务器配置为256GB或以上内存,这样可以有效帮助我们:
稳定处理大型单细胞数据集,避免在mapping或聚类步骤中因内存溢出导致中断;
明显加快数据处理速度,缩短每个样本的分析时间;
支持多任务并发运行,例如同时执行多个cellranger count任务或参考基因组构建(cellranger mkref)。
特别是当单次实验涉及5万细胞以上,或需要处理**高通量测序数据(如NovaSeq平台)**时,充足的内存资源尤为重要。
非科班医学生如何一步步部署生信服务器?
了解了硬件要求,接下来进入最实战的部分——生信服务器的部署步骤
第一步:选择合适的服务器购买或租用
根据实际需求不同,选择服务器的方式也有所不同:
个人科研项目:推荐购买性价比高的塔式工作站服务器,如戴尔T系列、联想ThinkStation系列,兼顾性能和成本。
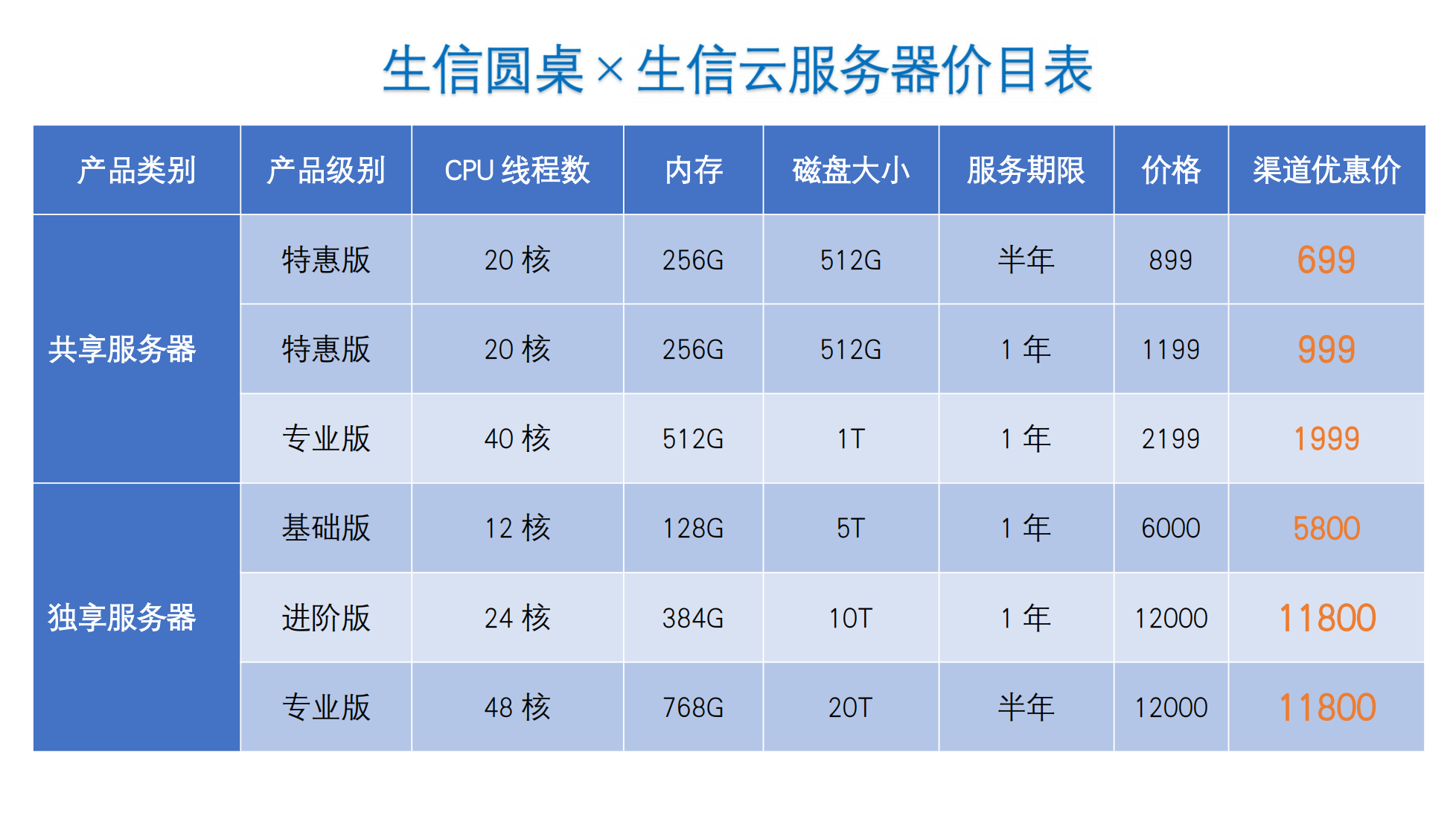
团队项目/重度使用:建议选择生物信息学专用云服务器。这类云服务器通常预装了生信分析环境(如1000+ R包),开箱即用,省去复杂运维,同时费用也比自建物理服务器更具性价比。
选择云服务器不仅可以省去前期大额投入,还能根据项目进展灵活调整配置,避免资源浪费。
第二步:安装Linux操作系统(推荐Ubuntu)
Cell Ranger官方支持的系统环境主要是Linux。对于非科班医学生而言,Ubuntu 20.04 LTS是最友好且资料丰富的选择。
如果你购买的是裸机服务器,基本流程如下:
从Ubuntu官网下载官方镜像文件;
使用Rufus等工具制作启动盘;
引导服务器,安装Ubuntu系统;
完成后配置SSH远程登录,方便后续操作与管理。
注意:如果你选用的是生信云服务器,这一步可以直接跳过,通常系统已经预装并优化好。
第三步:部署Cell Ranger运行环境
服务器系统安装完成后,需要搭建Cell Ranger分析环境。主要流程包括:
安装依赖库(如glibc、zlib等必要环境);
从10x Genomics官网下载最新版Cell Ranger软件;
解压文件,并配置环境变量(编辑.bashrc添加路径);
测试安装是否成功(运行命令cellranger mkfastq --help查看帮助信息)。
此外,如果计划分析自己的数据,还需提前准备参考基因组(reference genome),可以使用cellranger mkref命令生成对应的参考库。
第四步:基本使用技巧与维护建议
为了保证服务器长时间稳定运行,掌握一些常用小技巧非常有必要:
任务提交:使用nohup命令让任务在后台运行,避免意外中断;
资源监控:安装htop实时监控CPU、内存使用情况,及时发现潜在瓶颈;
数据备份:分析结果及时备份至云存储盘或外接硬盘,防止数据丢失带来无法挽回的损失。
这些小细节,往往能在长时间科研项目中极大提高整体效率与稳定性。
总结:非科班医学生也能轻松搞定生信服务器!
部署一台能够流畅运行Cell Ranger的生信服务器,并不需要复杂的编程基础。只要根据本篇攻略,选对硬件、装好系统、配置好环境,即使是零基础的非科班医学生,也能轻松完成部署,跑起自己的单细胞测序分析项目!
掌控了自己的服务器,就掌握了科研的节奏。
别再因为设备卡顿、环境搭建困难而影响进度——今天就动手搭建属于你的高效生信服务器吧!
👉 如果你希望获取详细配置清单、实用安装脚本或者生信服务器部署一键包,欢迎在评论区留言,我可以免费分享哦!