一.说明
monocle3 是一个用于分析单细胞转录组数据的 R 包。它提供了强大的工具来进行细胞轨迹分析、细胞类型标注以及其他高级单细胞数据分析功能。下面是如何在你的系统中安装 monocle3 包的详细步骤
二.安装(有时比较慢,需要耐心等待)
作者使用的是西柚云生信服务器,系统版本是Ubuntu 20.04版本。安装需要依次执行下面命令。
在linux系统上命令行执行的:
sudo add-apt-repository ppa:marutter/c2d4u4.0+
sudo add-apt-repository ppa:marutter/rrutter4.0
sudo apt update
sudo apt-get install jags
sudo apt-get install gdal-bin libgdal-dev在R里面执行的
R
remotes::install_github("cole-trapnell-lab/monocle3")验证安装
安装完成后,你可以通过加载 monocle3 包来验证安装是否成功。在 R 控制台中运行:
R
复制代码
library(monocle3)如果没有错误提示,那么安装成功。
三.常见问题
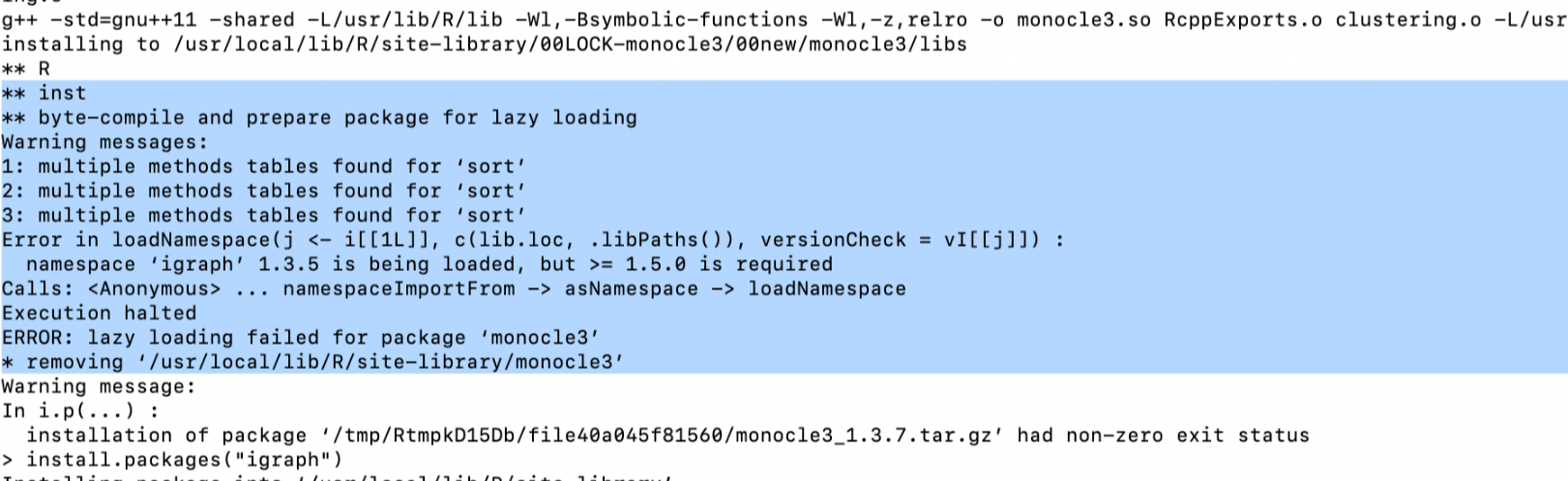
monocle安装过程中经常会需要安装其他依赖的R包,如果出现了报错可以一次解决并重新安装monocle。比如咱们图上的报错信息,提示说igraph的版本需要升级。那么我们只需要先安装igraph 再重新执行 remotes::install_github("cole-trapnell-lab/monocle3")
install.packages("igraph")
remotes::install_github("cole-trapnell-lab/monocle3") 安装过程中遇到错误:确保你的 R 版本是最新的,并且你的网络连接正常。有时错误可能是由于依赖项的版本不兼容或网络问题引起的。
加载包时出现错误:检查是否有其他包依赖未安装,或者尝试重新安装
monocle3包。BiocManager 安装失败:确保你的 R 版本符合 BiocManager 的要求,通常 R 4.0.0 及以上版本是支持的。
如果安装过程报错sf这个包问题,也可以看一下我对这个包的安装分享:安装install.packages("sf")报错解决办法.checking GDAL: checking whether PROJ | 生信圆桌 (tebteb.cc)
四.结论
安装 monocle3 包是一个简单的过程,只需要几个步骤即可完成但是需要准备一些环境。一旦安装成功,你就可以开始使用 monocle3 进行单细胞数据分析了。如果你在安装过程中遇到任何问题,可以查阅相关文档或寻求社区支持。
希望这篇博客对你有所帮助!如果你需要视频教程帮助,我们也帮你准备了视频版。
视频教程:
马上体验少走弯路,高效分析;了解生信云,访问 【生信圆桌x生信专用云服务器】 : www.tebteb.cc
注册下单,填写优惠码【yflalalaa】立减200
